通过PAML中的CODEML模块计算dnds的过程以及踩坑
最近帮女朋友做毕业设计的时候用到了 PAML这个软件的codeml功能,发现网上相关的资料很少,于是把自己踩的一些坑分享一下,希望能帮到其他有相同困难的人
一、下载与安装
PAML软件下载地址
http://abacus.gene.ucl.ac.uk/software/paml4.9j.tgz
DAMBE软件下载地址
http://dambe.bio.uottawa.ca/DAMBE/dambe_install_win.aspx
二、使用方法
首先准备好你的fas文件
我们需要将fas文件转换一下格式,方法很多,我这边说两种方法,这两种方法最后得到的文件内容完全相同,只是后缀名不同
方法一:
使用python脚本转换
将你的*.fas文件与脚本放在同一目录下,执行脚本,会生成一个.phy文件
import re
with open('seven.fas', 'r') as fin:
sequences = [(m.group(), ''.join(m.group().split()))
for m in re.finditer(r'(?m)^>([^ \n]+)[^\n]*([^>]*)', fin.read())]
with open('seven.phy', 'w') as fout:
fout.write('%d %d\n' % (len(sequences), len(sequences[][])))
for item in sequences:
fout.write('%-20s %s\n' % item)
方法二:
使用DAMBE软件转换格式
1.打开DAMBE,选择 File -> Open standard sequence file -> 文件类型选择为包含 fas 类型 -> 选择你的fas文件
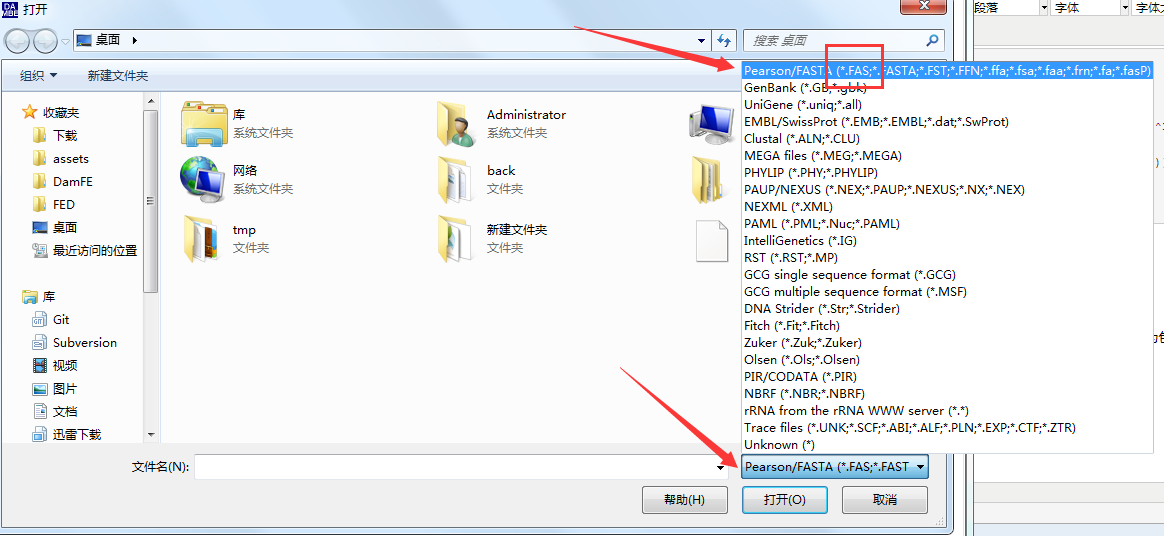
2.点击 go

3.点击 File -> save or convert sequence format -> 选择 paml 格式
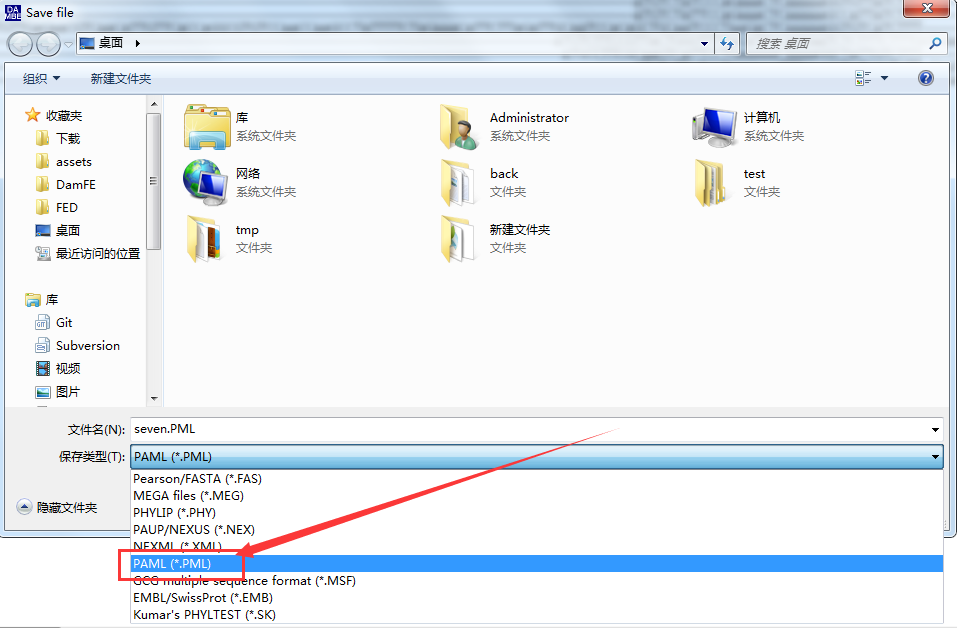
4.手动修改 *.pml 的后缀名为 *.nuc
通过以上两个方法会得到一份 *.phy 或者 *.nuc 文件
接下来需要去除序列中的终止密码子
你可以全选文件内容查找替换 将 TAG/TAA/TGA 替换为 ---
也可以使用下面这个python脚本
import re with open(r'seven.phy', 'r') as f:
content = f.read()
content = content.replace("TAG","---")
content = content.replace("TAA", "---")
content = content.replace("TGA", "---")
# print(content) with open('sevenend.phy', 'w') as f:
f.write(content)
会生成一个去除过终止密码子的文件
现在将这个处理过后的序列文件*.phy与树文件、配置文件codeml.ctl三个放在 \paml4.9j\bin 目录下
配置文件codeml.ctl内容如下可参考 一般修改前面三行即可 按顺序为序列文件名 树文件名 输出文件名
seqfile = seven.nuc * sequence data filename
treefile = Newick * tree structure file name
outfile = test.txt * main result file name noisy = * ,,,,: how much rubbish on the screen
verbose = * : concise; : detailed, : too much
runmode = - * : user tree; : semi-automatic; : automatic
* : StepwiseAddition; (,):PerturbationNNI; -: pairwise seqtype = * :codons; :AAs; :codons-->AAs
CodonFreq = * :/ each, :F1X4, :F3X4, :codon table * ndata =
clock = * :no clock, :clock; :local clock; :CombinedAnalysis
aaDist = * :equal, +:geometric; -:linear, -:G1974,Miyata,c,p,v,a
aaRatefile = dat/jones.dat * only used for aa seqs with model=empirical(_F)
* dayhoff.dat, jones.dat, wag.dat, mtmam.dat, or your own model =
* models for codons:
* :one, :b, : or more dN/dS ratios for branches
* models for AAs or codon-translated AAs:
* :poisson, :proportional, :Empirical, :Empirical+F
* :FromCodon, :AAClasses, :REVaa_0, :REVaa(nr=) NSsites = * :one w;:neutral;:selection; :discrete;:freqs;
* :gamma;:2gamma;:beta;:beta&w;:betaγ
* :beta&gamma+; :beta&normal>; :&2normal>;
* :3normal> icode = * :universal code; :mammalian mt; -:see below
Mgene =
* codon: :rates, :separate; :diff pi, :diff kapa, :all diff
* AA: :rates, :separate fix_kappa = * : kappa fixed, : kappa to be estimated
kappa = * initial or fixed kappa
fix_omega = * : omega or omega_1 fixed, : estimate
omega = . * initial or fixed omega, for codons or codon-based AAs fix_alpha = * : estimate gamma shape parameter; : fix it at alpha
alpha = . * initial or fixed alpha, :infinity (constant rate)
Malpha = * different alphas for genes
ncatG = * # of categories in dG of NSsites models getSE = * : don't want them, 1: want S.E.s of estimates
RateAncestor = * (,,): rates (alpha>) or ancestral states ( or ) Small_Diff = .5e-
cleandata = * remove sites with ambiguity data (:yes, :no)?
* fix_blength = * : ignore, -: random, : initial, : fixed, : proportional
method = * Optimization method : simultaneous; : one branch a time * Genetic codes: :universal, :mammalian mt., :yeast mt., :mold mt.,
* : invertebrate mt., : ciliate nuclear, : echinoderm mt.,
* : euplotid mt., : alternative yeast nu. : ascidian mt.,
* : blepharisma nu.
* These codes correspond to transl_table to of GENEBANK.
在此目录下打开命令行
输入一下命令即可
codeml
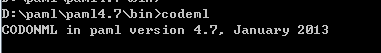
当前目录下就会出现结果文件 test.txt 以及其他文件了
在过程中我遇到过许多报错提供给大家参考一下
67 columns are converted into ??? because of stop codons
这个报错是因为没有去除文件中的终止密码子,可以参考上面的步骤去除
Error: Error in sequence data file: . in 1st seq.?.
Error: check #seqs and tree: perhaps too many '('?.
Make sure to separate the sequence from its name by 2 or more spaces.
以上报错均为你的序列文件内容/格式有问题,麻烦按照上面的步骤重新生成序列文件或者参考其他人的文件格式
最新文章
- PHP 正则表达式 修饰符
- 【Java EE 学习 74 上】【数据采集系统第六天】【使用Jfreechart的统计图实现】【Jfreechart的基本使用方法】
- 【翻译svg教程 】svg 的坐标系统
- 5天玩转C#并行和多线程编程 —— 第三天 认识和使用Task
- python 代码片段23
- flask-sqlalchemy分表解决方案
- 使用apt-fast 来加速你的Ubuntu 的apt
- Javascript基础(2)
- FlexSlider是一个非常出色的jQuery滑动切换插件
- Oracle EBS-SQL (CST-1):检查BOM历史成本查询(Average Cost).sql
- Django操作model时刻,一个错误:AttributeError:’ProgrammingError’ object has no attribute ‘__traceback__’
- leetcode70
- Access中的自定义排序设置方式
- Python3 的序列
- 多线程学习笔记-深入理解ThreadPoolExecutor
- win10 开机自启指定软件
- java 两个List集合各种情况对比处理
- 【Vue】谈Vue的依赖追踪系统 ——搞懂methods watch和compute的区别和联系
- JS如何判断一个对象是否为空、是否有某个属性
- PyCharm+Miniconda3安装配置教程